PLATAFORMAS BIOSENSORAS ELECTROANALÍTICAS PARA LA DETERMINACIÓN DE METILACIONES EN ÁCIDOS NUCLEICOS A NIVEL GLOBAL Y REGIONAL
|
Descripción |
Las tecnologías desarrolladas permiten la determinación individual o simultánea de las principales bases metiladas en ADN (5-metilcitosina, 5-mC y 5-hidroximetilcitosina, 5-hmC) y ARN (N6-metiladenosina, m6A), tanto a nivel global como regional, de forma rápida, fiable y sencilla. Las metodologías puestas a punto se basan en el empleo de biosensores amperométricos que utilizan soportes micromagnéticos (MBs) funcionalizados con bioreceptores adecuados y detección amperométrica sobre electrodos serigrafiados desechables.
En estos biodispositivos, la señal amperométrica obtenida, que puede relacionarse con la cantidad de base metilada diana, se basa en las reacciones enzimáticas y electroquímicas implicadas en el sistema peroxidasa de rábano (HRP)/hidroquinona (HQ)/H2O2 que tienen lugar tras la captura de las MBs en la superficie de un transductor electroquímico desechable a un potencial constante apropiado.
|
¿Cómo funciona? |
Las tecnologías se basan en el empleo de MBs modificadas con una secuencia sintética de ADN complementaria a la región del gen donde se quiere detectar la metilación, para su determinación a nivel regional (Figura 1a), o con anticuerpos comerciales selectivos a la base metilada que se quiere determinar, para su determinación a nivel global (Figura 1b).
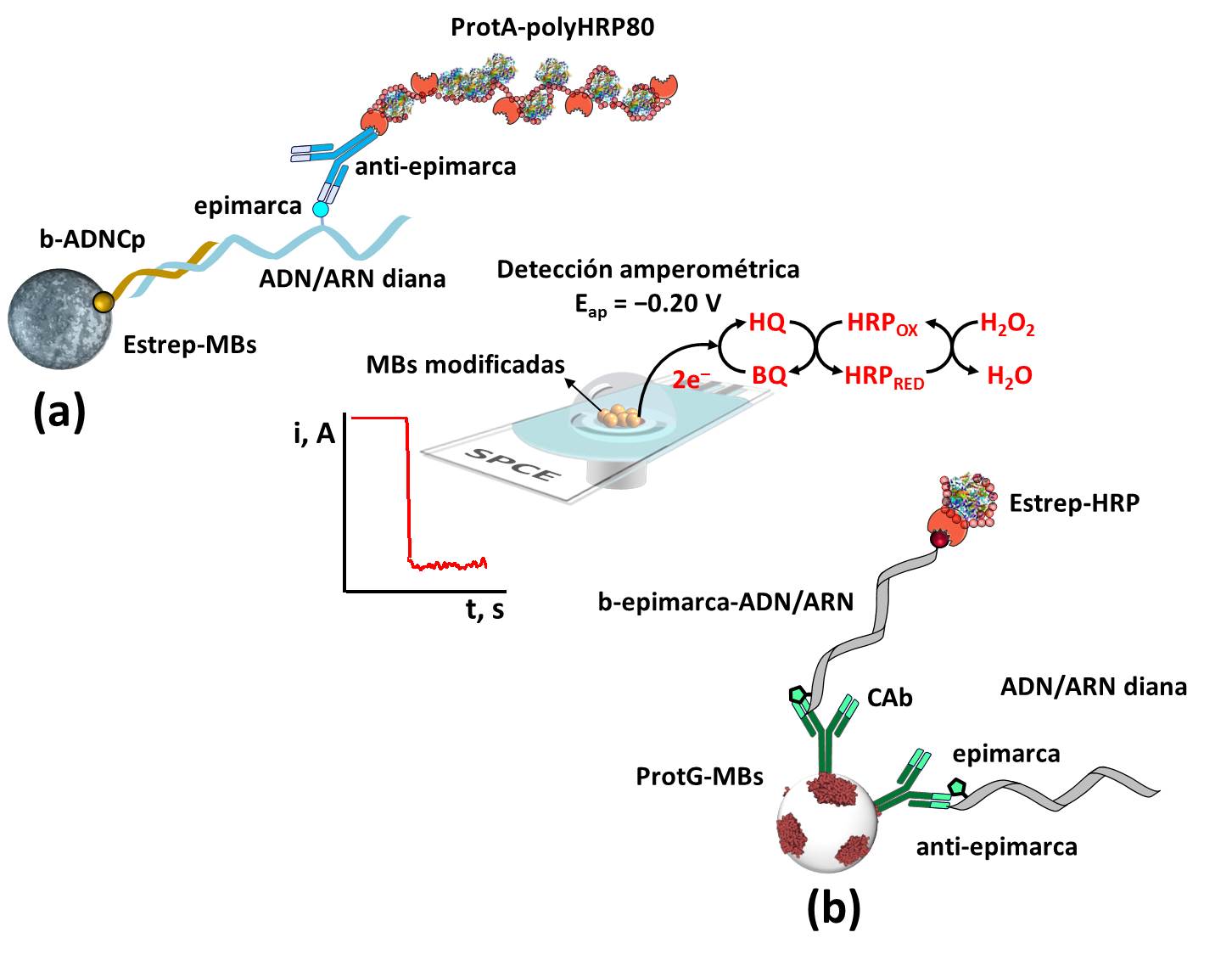
Figura 1. Diagrama esquemático de las plataformas biosensoras desarrolladas para la determinación de metilaciones en ácidos nucleicos a nivel regional (a) y global (b).
Para la determinación a nivel regional (Figura 1a), el ADN o ARN metilado diana se captura selectivamente sobre MBs modificadas con una sonda sintética de ADN biotinilada que hibrida con la región adyacente a la región donde se quiere evaluar la presencia de metilaciones. A continuación, el ácido nucleico metilado se detecta empleando un anticuerpo selectivo a la base metilada diana que se marca enzimáticamente con HRP empleando bioreceptores disponibles comercialmente con múltiples moléculas de HRP (Proteína A-polyHRP80).
Para la determinación a nivel global (Figura 1b), se emplean formatos de inmunoensayo de tipo competitivo directo sobre los inmunoconjugados en los que el ADN o ARN diana metilado compite por los limitados sitios de unión de los anticuerpos inmovilizados sobre las MBs con un oligómero de ADN o ARN biotinilado sintético que porta una única base metilada y que se marca con la enzima HRP empleando el reactivo comercial adecuado (conjugado de estreptavidina-HRP).
En todos los casos, las MBs modificadas se capturan magnéticamente sobre la superficie de electrodos (o arrays de electrodos) desechables y se realiza la transducción amperométrica empleando el sistema HRP/ HQ/H2O2 obteniéndose una variación de corriente catódica atribuida a la reducción enzimática del H2O2 mediada por la HQ, que puede relacionarse con la concentración de la base metilada diana.
|
Ventajas |
Entre las ventajas competitivas de las metodologías desarrolladas destacan:
- Fácilmente trasladables a la determinación de otras epimarcas de interés.
- Por el tipo de detección e instrumentación que emplean, sencilla implementación en dispositivos de diagnóstico en el punto de atención (POC) o de tipo “muestra-resultado”.
- Posibilidad de determinación simultánea de 5-mC y 5-hmC, las dos bases metiladas más frecuentes y similares en ADN.
- Sensibilidad a nivel de una única base sin necesidad de emplear etapas de amplificación de ácidos nucleicos.
- Capaces de operar a temperatura ambiente.
- Proporcionar resultados cuantitativos en tiempos inferiores a 2 h y con coste muy asequible (< 2 €/determinación).
|
¿Dónde se ha desarrollado? |
Estas bioplataformas han sido desarrolladas en el Grupo de Electroanálisis y (Bio)sensores Electroquímicos de la Facultad de Ciencias Químicas de la Universidad Complutense de Madrid (Grupo GEBE, Grupo UCM Referencia 910319) y en colaboración con investigadores y clínicos del Instituto de Salud Carlos III y el Hospital Universitario La Paz.
|
Y además |
Los resultados obtenidos confirman unas excelentes prestaciones para realizar determinaciones fiables en células, biopsias humanas de distinta naturaleza (sólidas y líquidas) y directamente en suero humano sin extracción previa de ADN genómico.
Se ha confirmado, asimismo, el potencial de estas bioplataformas para discriminar entre individuos sanos y afectados por cáncer mediante la determinación del nivel global de 5-mC y 5-hmC en ADN genómico extraído de los tejidos (sensibilidad y especificidad del 89 % y 67 %, respectivamente) o mediante la detección de 5-mC a nivel regional directamente en el suero sin extracción previa de material genético. Además, permiten caracterizar la agresividad de las células cancerosas mediante la detección de m6A a nivel global en una pequeña cantidad de ARN total extraído sin necesidad de enriquecimiento o fragmentación. En las determinaciones en células y tejidos humanos, la cantidad de ADN genómico o de ARN celular empleado por determinación está comprendida entre 10 y 100 ng.
La incorporación de estas bioherramientas, que proporcionan resultados consistentes con los suministrados por las metodologías convencionales (RT-PCR), al análisis clínico rutinario conllevaría ventajas importantes en términos de sencillez, rapidez, coste, portabilidad y posibilidad de multiplexado, fiabilidad para el diagnóstico temprano y el pronóstico de enfermedades prevalentes como el cáncer y mejora de la eficiencia de la terapia aplicada.
Página web con información adicional: https://gebeucm.wordpress.com/
|
Contacto |
|
© Oficina de Transferencia de Resultados de Investigación – UCM |
|
Descargas PDF |
|
Clasificación |
|
Investigadores responsables |
Susana Campuzano Ruiz: susanacr@quim.ucm.es
María Pedrero Muñoz: mpedrero@quim.ucm.es
José M. Pingarrón Carrazón: pingarro@quim.ucm.es
Departamento: Química Analítica
Facultad: Ciencias Químicas